Евгений Кунин - Логика случая. О природе и происхождении биологической эволюции
- Название:Логика случая. О природе и происхождении биологической эволюции
- Автор:
- Жанр:
- Издательство:Литагент «Центрполиграф»a8b439f2-3900-11e0-8c7e-ec5afce481d9
- Год:2014
- Город:Москва
- ISBN:978-5-227-04982-7
- Рейтинг:
- Избранное:Добавить в избранное
-
Отзывы:
-
Ваша оценка:
Евгений Кунин - Логика случая. О природе и происхождении биологической эволюции краткое содержание
В этой амбициозной книге Евгений Кунин освещает переплетение случайного и закономерного, лежащих в основе самой сути жизни. В попытке достичь более глубокого понимания взаимного влияния случайности и необходимости, двигающих вперед биологическую эволюцию, Кунин сводит воедино новые данные и концепции, намечая при этом дорогу, ведущую за пределы синтетической теории эво люции. Он интерпретирует эволюцию как стохастический процесс, основанный на заранее непредвиденных обстоятельствах, ограниченный необходимостью поддержки клеточной организации и направляемый процессом адаптации. Для поддержки своих выводов он объединяет между собой множество концептуальных идей: сравнительную геномику, проливающую свет на предковые формы; новое понимание шаблонов, способов и непредсказуемости процесса эволюции; достижения в изучении экспрессии генов, распространенности белков и других фенотипических молекулярных характеристик; применение методов статистической физики для изучения генов и геномов и новый взгляд на вероятность самопроизвольного появления жизни, порождаемый современной космологией.
Логика случая демонстрирует, что то понимание эволюции, которое было выработано наукой XX века, является устаревшим и неполным, и обрисовывает фундаментально новый подход – вызывающий, иногда противоречивый, но всегда основанный на твердых научных знаниях.
Логика случая. О природе и происхождении биологической эволюции - читать онлайн бесплатно ознакомительный отрывок
Интервал:
Закладка:
Для появления примитивной системы сопряженной репликации-трансляции, что в данном контексте рассматривается как революционная стадия, требования гораздо жестче. Как минимум, необходимо спонтанное появление следующего:
• Две рРНК, с общим размером не менее 1000 нуклеотидов.
• Примерно 10 примитивных адаптеров по 30 нуклеотидов каждый, в целом около 300 нуклеотидов.
• По меньшей мере одна РНК, кодирующая репликазу, размером примерно 500 нуклеотидов (оценка снизу). В принятой модели, n = 1800, и в результате E < 10 –1018.
Другими словами, даже в нашей игрушечной модели, которая предполагает сильно преувеличенную скорость синтеза РНК, вероятность случайного зарождения системы трансляция – репликация в единственной Н-области будет P < 10 –1018. Очевидно, эта версия революционной стадии может рассматриваться только в контексте вселенной с бесконечным (или, по меньшей мере, очень большим) количеством Н-областей.
Модель, рассмотренная здесь, ни в коем случае не предполагалась реалистичной. Она только иллюстрирует разницу в требованиях, накладываемых на вероятность возникновения разных версий революционных систем, и следовательно, связь этой версии с разными космологическими моделями вселенной.
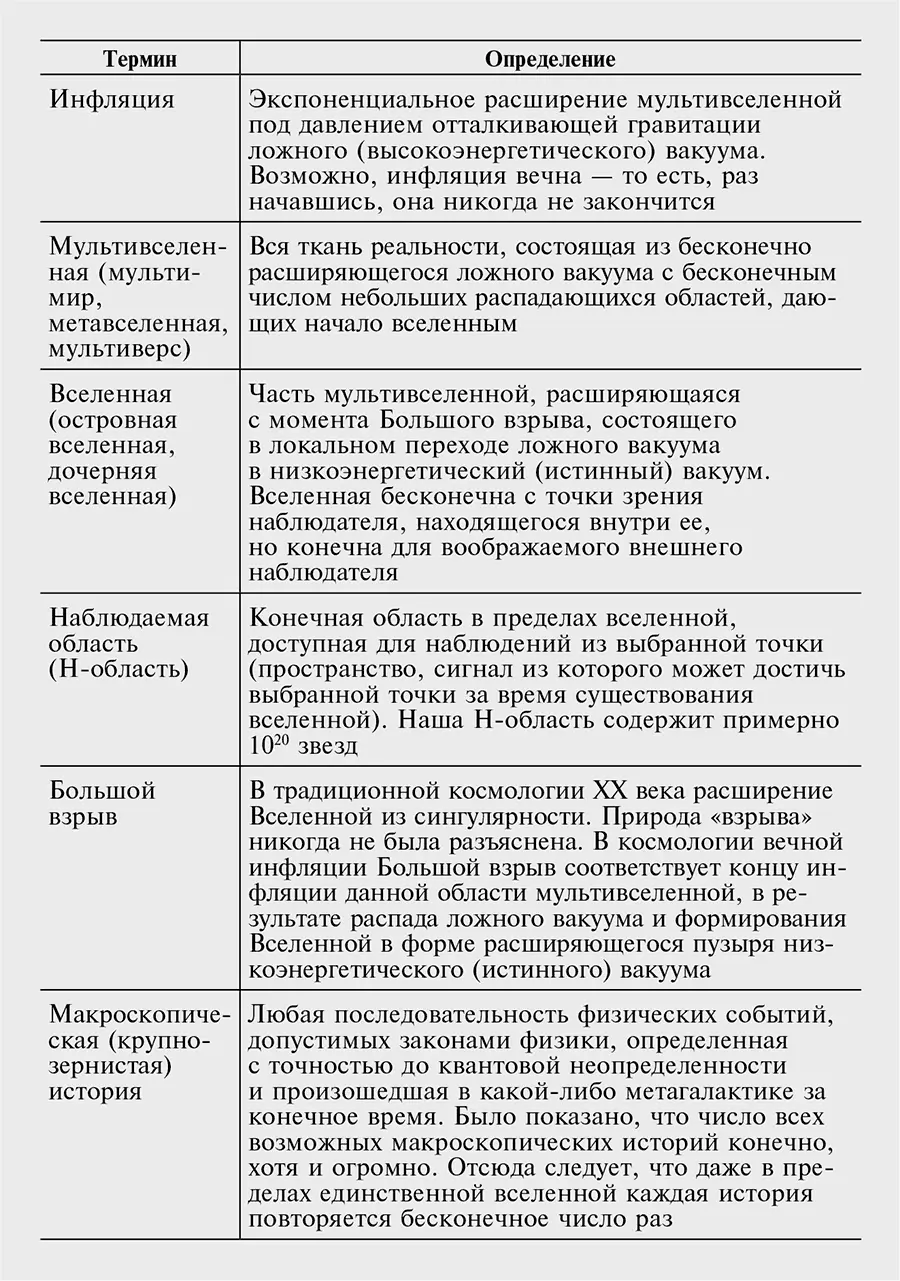
Таблица II-1. Новые определения и новые интерпретации известных определений в модели МММ.

Литература
(1948) O Polozhenii V Biologicheskoi Nauke. Stenograficheskii Otchet Sessii Vsesoyuznoi Akademii Selskohozyastvennyh Nauk Imeni V. I. Lenina. (On the Situation in Biological Science. A Transcript of the Session of the V. I. Lenin All-Union Academy of Agricultural Sciences, July 31-August 7, 1948. Moscow, USSR: The State Agricultural Literature Publishers.
‘t Hooft, G. (1993) Dimensional Reduction in Quantum Gravity. gr-qc/9310026.
Adami C. (2002) What Is Complexity? Bioessays 24: 1,085—1,094.
Adl, S. M., A. G. Simpson, M. A. Farmer, R. A. Andersen, O. R. Anderson, J. R. Barta, S. S. Bowser, G. Brugerolle, R. A. Fensome, S. Fredericq, T. Y. James, S. Karpov, P. Kugrens, J. Krug, C. E. Lane, L. A. Lewis, J. Lodge, D. H. Lynn, D. G. Mann, R. M. McCourt, L. Mendoza, O. Moestrup, S. E. Mozley-Standridge, T. A. Nerad, C. A. Shearer, A. V. Smirnov, F. W. Spiegel, and M. F. Taylor. (2005) The New Higher Level Classification of Eukaryotes with Emphasis on the Taxonomy of Protists. J Eukaryot Microbiol 52: 399–451.
Agol V. I. (1974) Towards the System of Viruses. Biosystems 6: 113–132.
Agol V. I. , and A. P. Gmyl. (2010) Viral Security Proteins: Counteracting Host Defences. Nat Rev Microbiol 8: 867–878.
Ahlquist P. (2006) Parallels among Positive-Strand RNA Viruses, Reverse-Transcribing Viruses and Double-Stranded RNA Viruses. Nat Rev Microbiol 4: 371–382.
Alic N., N. Ayoub, E. Landrieux, E. Favry, P. Baudouin-Cornu, M. Riva , and C. Carles. (2007) Selectivity and Proofreading Both Contribute Significantly to the Fidelity of RNA Polymerase III Transcription. Proc Natl Acad Sci USA 104: 10,400—10,405.
Allen, E. E. , and J. F. Banfield. (2005) Community Genomics in Microbial Ecology and Evolution. Nat Rev Microbiol 3: 489–498.
Alperovitch-Lavy, A., I. Sharon, F. Rohwer, E. M. Aro, F. Glaser, R. Milo, N. Nelson, and O. Beja. (2011) Reconstructing a Puzzle: Existence of Cyanophages Containing Both Photosystem-I and Photosystem-II Gene Suites Inferred from Oceanic Metagenomic Datasets. Environ Microbiol 13: 24–32.
Altschul, S. F., T. L. Madden, A. A. Schaffer, J. Zhang, Z. Zhang, W. Miller , and D. J. Lipman. (1997) Gapped Blast and Psi-Blast: A New Generation of Protein Database Search Programs. Nucleic Acids Res 25: 3,389—3,402.
Altstein, A. D. (1987) Origin of the Genetic System. Mol Biol 21: 309–322.
Amaral, P. P., M. E. Dinger, T. R. Mercer, and J. S. Mattick. (2008) The Eukaryotic Genome as an RNA Machine. Science 319: 1,787—1,789.
Anantharaman, V., E. V. Koonin, and L. Aravind. (2002) Comparative Genomics and Evolution of Proteins Involved in RNA Metabolism. Nucleic Acids Res 30: 1,427—1,464.
Anfinsen, C. B. (1973) Principles That Govern the Folding of Protein Chains. Science 181: 223–230.
Aquadro, C. F. (1997) Insights into the Evolutionary Process from Patterns of DNA Sequence Variability. Curr Opin Genet Dev 7: 835–840.
Aravind, L., V. Anantharaman, S. Balaji, M. M. Babu, and L. M. Iyer. (2005) The Many Faces of the Helix-Turn-Helix Domain: Transcription Regulation and Beyond. FEMS Microbiol Rev 29: 231–262.
Aravind, L., L. M. Iyer and E. V. Koonin. (2006) Comparative Genomics and Structural Biology of the Molecular Innovations of Eukaryotes. Curr Opin Struct Biol 16: 409–419.
Aravind, L., and E. V. Koonin. (1999) DNA-Binding Proteins and Evolution of Transcription Regulation in the Archaea. Nucleic Acids Res 27: 4,658—4,670.
Aravind, L., and E. V. Koonin. (2001) The DNA-Repair Protein Alkb, Egl-9, and Leprecan Define New Families of 2-Oxoglutarate – and Iron-Dependent Dioxygenases. Genome Biol 2: RESEARCH0007.
Aravind, L., R. Mazumder, S. Vasudevan, and E. V. Koonin. (2002) Trends in Protein Evolution Inferred from Sequence and Structure Analysis. Curr Opin Struct Biol 12: 392–399.
Aravind, L., R. L. Tatusov, Y. I. Wolf, D. R. Walker, and E. V. Koonin. (1998) Evidence for Massive Gene Exchange Between Archaeal and Bacterial Hyperthermophiles. Trends Genet 14: 442–444.
Aravind, L., D. R. Walker, and E. V. Koonin. (1999) Conserved Domains in DNA Repair Proteins and Evolution of Repair Systems. Nucleic Acids Res 27: 1,223—1,242.
Assis, R., and A. S. Kondrashov. (2009) Rapid Repetitive Element-Mediated Expansion of piRNA Clusters in Mammalian Evolution. Proc Natl Acad Sci USA 106: 7,079—7,082.
Assis, R., A. S. Kondrashov, E. V. Koonin, and F. A. Kondrashov. (2008) Nested Genes and Increasing Organizational Complexity of Metazoan Genomes. Trends Genet 24: 475–478.
Atkins, J. F., R. F. Gesteland, and T. R. Cech, eds. (2010) RNA Worlds: From Life’s Origins to Diversity in Gene Regulation. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
Auster, P. (1991) The Music of Chance . New York: Penguin.
Autexier, C., and N. F. Lue. (2006) The Structure and Function of Telomerase Reverse Transcriptase. Annu Rev Biochem 75: 493–517.
Baaske, P., F. M. Weinert, S. Duhr, K. H. Lemke, M. J. Russell, and D. Braun. (2007) Extreme Accumulation of Nucleotides in Simulated Hydrothermal Pore Systems. Proc Natl Acad Sci USA 104: 9,346—9,351.
Babu, M., N. Beloglazova, R. Flick, C. Graham, T. Skarina, B. Nocek, A. Gagarinova, O. Pogoutse, G. Brown, A. Binkowski, S. Phanse, A. Joachimiak, E. V. Koonin, A. Savchenko, A. Emili, J. Greenblatt, A. M. Edwards, and A. F. Yakunin. (2011) A Dual Function of the CRISPR-Cas System in Bacterial Antivirus Immunity and DNA Repair. Mol Microbiol 79: 484–502.
Ball, P. (2011) A Metaphor Too Far. Nature doi:10.1038/news.2011.115.
Bailey, K. A., F. Marc, K. Sandman, and J. N. Reeve. (2002) Both DNA and His-tone Fold Sequences Contribute to Archaeal Nucleosome Stability. J Biol Chem 15: 9293–9301.
Baltimore, D. (1971) Expression of Animal Virus Genomes. Bacteriol Rev 35: 235–241.
Bangham, C. R., and T. B. Kirkwood. (1993) Defective Interfering Particles and Virus Evolution. Trends Microbiol 1: 260–264.
Barabasi, A. L. (2002) Linked: The New Science of Networks . New York: Perseus Press.
Barabasi, A. L., and Z. N. Oltvai. (2004) Network Biology: Understanding the Cell’s Functional Organization. Nat Rev Genet 5: 101–113.
Barbrook, A. C., C. J. Howe, D. P. Kurniawan, and S. J. Tarr. (2010) Organization and Expression of Organellar Genomes. Philos Trans R Soc Lond B Biol Sci 365: 785–797.
Baross, J. A., and S. A. Hoffman. (1985) Submarine Hydrothermal Vents and Associated Gradient Environments As Sites for the Origin and Evolution of Life. Origins of Life 15: 327–345.
Barrangou, R., C. Fremaux, H. Deveau, M. Richards, P. Boyaval, S. Moineau, D. A. Romero, and P. Horvath. (2007) CRISPR Provides Acquired Resistance Against Viruses in Prokaryotes. Science 315: 1,709—1,712.
Barrick, J. E., D. S. Yu, S. H. Yoon, H. Jeong, T. K. Oh, D. Schneider, R. E. Lenski, and J. F. Kim. (2009) Genome Evolution and Adaptation in a Long-Term Experiment with Escherichia coli. Nature 461: 1243–1247.
Barrow, J. D., and F. J. Tipler. (1988) The Anthropic Cosmological Principle . Oxford: Oxford University Press.
Barrowman, J., D. Bhandari, K. Reinisch, and S. Ferro-Novick. (2010) Trapp Complexes in Membrane Traffic: Convergence Through a Common Rab. Nat Rev Mol Cell Biol 11: 759–763.
Barton, N. H. (2000) Genetic Hitchhiking. Philos Trans R Soc Lond B Biol Sci 355: 1,553—1,562.
Barton, N. H., and J. B. Coe. (2009) On the Application of Statistical Physics to Evolutionary Biology. J Theor Biol 259: 317–324.
Basu, M. K., E. Poliakov, and I. B. Rogozin. (2009) Domain Mobility in Proteins: Functional and Evolutionary Implications. Brief Bioinform 10: 205–216.
Beardmore, R. E., I. Gudelj, D. A. Lipson, and L. D. Hurst. (2011) Metabolic Trade-Offs and the Maintenance of the Fittest and the Flattest. Nature [Epub ahead of print].
Beeby, M., B. D. O’Connor, C. Ryttersgaard, D. R. Boutz, L. J. Perry, and T. O. Yeates. (2005) The Genomics of Disulfide Bonding and Protein Stabilization in Thermophiles. PLoS Biol 3: e309.
Читать дальшеИнтервал:
Закладка:









