Денис Соломатин - Математические модели в естественнонаучном образовании. Том II
- Название:Математические модели в естественнонаучном образовании. Том II
- Автор:
- Жанр:
- Издательство:неизвестно
- Год:2022
- ISBN:нет данных
- Рейтинг:
- Избранное:Добавить в избранное
-
Отзывы:
-
Ваша оценка:
Денис Соломатин - Математические модели в естественнонаучном образовании. Том II краткое содержание
Математические модели в естественнонаучном образовании. Том II - читать онлайн бесплатно ознакомительный отрывок
Интервал:
Закладка:
Обратите внимание, что предположение о молекулярных часах неявно присутствовала в UPGMA. В примере, когда поместили 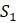 и
и 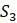 на концы ветвей одинаковой длины, предположили, что количество мутаций, которые каждый из них претерпел от своего общего предка, было одинаковым. Метод UPGMA всегда размещает все таксоны на одинаковом расстоянии от корня, так что количество мутаций от корня до любого таксона одинаково.
на концы ветвей одинаковой длины, предположили, что количество мутаций, которые каждый из них претерпел от своего общего предка, было одинаковым. Метод UPGMA всегда размещает все таксоны на одинаковом расстоянии от корня, так что количество мутаций от корня до любого таксона одинаково.
Вторым рассмотрим алгоритм Фитча-Марголиаша. Этот метод немного сложнее, чем UPGMA, но основан на том же подходе. Тем не менее, попытаемся отказаться от предположения UPGMA о молекулярных часах.
Прежде чем изложить алгоритм, сделаем несколько математических наблюдений. Во-первых, если попытаемся поместить 3 таксона на некорневое дерево, то будет только одна топология, которую необходимо учитывать. Кроме того, для 3 таксонов можем назначить желаемые длины ребер, чтобы точно соответствовать данным. Чтобы убедиться в этом, рассмотрим дерево на рисунке 5.9. Если есть некоторые данные о расстоянии 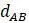 ,
, 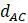 и
и 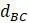 , то можно составить систему уравнений
, то можно составить систему уравнений 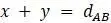 ,
, 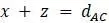 ,
, 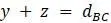 .
.
Эти уравнения могут быть решены либо путем записи системы в виде матричного уравнения и нахождения обратной матрицы, либо путем подстановки формулы для одной переменной, полученной из одного уравнения, в другие. Любой способ гарантированно приведёт к следующему решению 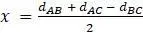 ,
, 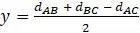 ,
, 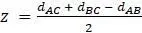 .
.
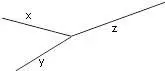
Рисунок 5.9. Некорневое 3-таксонное дерево.
Будем называть эти формулы 3-точечными формулами для подгонки таксонов к дереву. К сожалению, с более чем 3 таксонами точная подгонка данных к дереву обычно невозможна. Однако алгоритм Фитча-Марголиаша (кратко называемый в таблицах как FM) использует случай 3 таксонов для обработки большего количества таксонов. Теперь объясним работу алгоритма на примере. Будем использовать данные о расстоянии, приведенные в таблице 5.4.
Таблица 5.4. Расстояния между таксонами
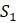
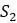
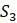
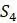
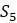
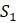
.31 1.01 .75 1.03
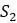
1.00 .69 .90
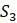
.61 .42
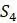
.37
Начинаем с выбора ближайшей пары таксонов для присоединения, как это делали в UPGMA. Глядя на таблицу расстояний, 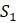 и
и 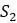 являются первой парой, которая соединится. Чтобы соединить их, не помещая их на равное расстояние от общего предка, временно сводим задачу к случаю 3-таксонов, объединяя все остальные таксоны в группу. Таким образом, для имеющихся данных вводим группу
являются первой парой, которая соединится. Чтобы соединить их, не помещая их на равное расстояние от общего предка, временно сводим задачу к случаю 3-таксонов, объединяя все остальные таксоны в группу. Таким образом, для имеющихся данных вводим группу 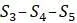 . Находим расстояние от каждого из
. Находим расстояние от каждого из 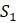 и
и 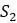 до группы, усредняя их расстояния до каждого члена группы. Таким образом, расстояние от
до группы, усредняя их расстояния до каждого члена группы. Таким образом, расстояние от 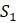 до
до 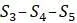 равно
равно 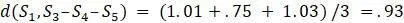 , в то время как от
, в то время как от 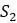 до
до 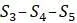 оно равно
оно равно 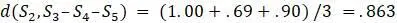 . Это дает таблицу 5.5.
. Это дает таблицу 5.5.
Таблица 5.5. Расстояния между группами; FM-алгоритм, шаг 1a
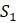
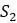
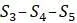
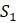
.31 .93
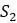
.863
Имея только три таксона в этой таблице, можем точно подогнать данные к дереву, используя 3-точечные формулы, чтобы получить рисунок 5.10. Ключевым моментом здесь является то, что 3-точечные формулы, в отличие от UPGMA, могут давать неравные расстояния таксонов от общего предка.

Рисунок 5.10. FM-алгоритм; шаг 1.
Теперь оставляем только ребра, заканчивающиеся в 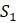 и
и 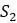 на рисунке 5.10, и возвращаемся к исходным данным. Помните, что группа
на рисунке 5.10, и возвращаемся к исходным данным. Помните, что группа 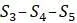 была нужна только временно, чтобы могли использовать 3-точечные формулы; пока не собирались объединять эти таксоны. Однако, поскольку объединили
была нужна только временно, чтобы могли использовать 3-точечные формулы; пока не собирались объединять эти таксоны. Однако, поскольку объединили 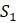 и
и 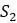 , объединяем их в группу для остальной части алгоритма, как сделали бы с UPGMA. Это формирует таблицу 5.6.
, объединяем их в группу для остальной части алгоритма, как сделали бы с UPGMA. Это формирует таблицу 5.6.
Таблица 5.6. Расстояния между группами; FM-алгоритм, шаг 1b
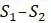
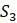
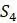
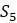
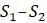
1.005 .72 .965
Читать дальшеИнтервал:
Закладка:










